当前位置:
X-MOL 学术
›
Water Res.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
The hospital sink drain biofilm resistome is independent of the corresponding microbiota, the environment and disinfection measures
Water Research ( IF 11.4 ) Pub Date : 2025-06-01 , DOI: 10.1016/j.watres.2025.123902
Aurélie Hennebique, Jostin Monge-Ruiz, Morgane Roger-Margueritat, Patrice Morand, Claire Terreaux-Masson, Max Maurin, Corinne Mercier, Caroline Landelle, Elena Buelow
Water Research ( IF 11.4 ) Pub Date : 2025-06-01 , DOI: 10.1016/j.watres.2025.123902
Aurélie Hennebique, Jostin Monge-Ruiz, Morgane Roger-Margueritat, Patrice Morand, Claire Terreaux-Masson, Max Maurin, Corinne Mercier, Caroline Landelle, Elena Buelow
|
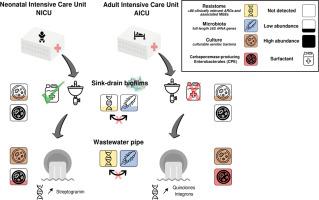
In hospitals, the transmission of antibiotic-resistant bacteria (ARB) may occur via biofilms present in sink drains, which can lead to infections. Despite the potential role of sink drains in the transmission of ARB in nosocomial infections, routine surveillance of these drains is lacking in most hospitals. As a result, there is currently no comprehensive understanding of the transmission of ARB and the dissemination of antimicrobial resistance genes (ARGs) and associated mobile genetic elements (MGEs) via sink drains. This study employed a multifaceted approach to monitor the total aerobic bacteria as well as the presence of carbapenemase-producing Enterobacterales (CPEs), the microbiota and the resistome of sink drain biofilms (SDBs) and hospital wastewater (WW) of two separate intensive care units (ICUs) in the same healthcare facility in France. Samples of SDB and WW were collected on a monthly basis, from January to April 2023, in the neonatal (NICU) and the adult (AICU) ICUs of Grenoble Alpes University Hospital. In the NICU, sink drain disinfection with surfactants was performed routinely. In the AICU, routine disinfection is not carried out. Culturable aerobic bacteria were quantified on non-selective media, and CPEs were screened using two selective agars. Isolates were identified by MALDI-TOF MS, and antibiotic susceptibility testing (AST) was performed on Enterobacterales and P. aeruginosa. The resistome was analyzed by high-throughput qPCR targeting >80 ARGs and MGEs. The overall bacterial microbiota was assessed via full-length 16S rRNA sequencing. No CPEs were isolated from SDBs in either ICU by bacterial culture. Culture-independent approaches revealed an overall distinct microbiota composition of the SDBs in the two ICUs. The AICU SDBs were dominated by pathogens containing Gram-negative bacterial genera including Pseudomonas, Stenotrophomona, Klebsiella, and Gram-positive Staphylococcus, while the NICU SDBs were dominated by the Gram-negative genera Achromobacter, Serratia, and Acidovorax, as well as the Gram-positive genera Weisella and Lactiplantibacillus. In contrast, the resistome of the SDBs exhibited no significant differences between the two ICUs, indicating that the abundance of ARGs and MGEs is independent of microbiota composition and disinfection practices. The AICU WW exhibited more distinct aerobic bacteria than the NICU WW. In addition, the AICU WW yielded 15 CPEs, whereas the NICU WW yielded a single CPE. All the CPEs were characterized at the species level. The microbiota of the NICU and AICU WW samples differed from their respective SDBs and exhibited distinct variations over the four-month period:the AICU WW contained a greater number of genes conferring resistance to quinolones and integron integrase genes, whereas the NICU WW exhibited a higher abundance of streptogramin resistance genes. Our study demonstrated that the resistome of the hospital SDBs in the two ICUs of the investigated healthcare institute is independent of the microbiota, the environment, and the local disinfection measures. However, the prevalence of CPEs in the WW pipes collecting the waste from the investigated drains differed. These findings offer valuable insights into the resilience of resistance genes in SDBs in ICUs, underscoring the necessity for innovative strategies to combat antimicrobial resistance in clinical environments.
中文翻译:

医院水槽排水生物膜抵抗组独立于相应的微生物群、环境和消毒措施
在医院,抗生素耐药菌 (ARB) 可能通过水槽排水管中的生物膜传播,这可能导致感染。尽管汇引流管在院内感染中 ARB 的传播中具有潜在作用,但大多数医院缺乏对这些引流管的常规监测。因此,目前对 ARB 的传播以及抗菌素耐药基因 (ARG) 和相关的移动遗传元件 (MGE) 通过下水道的传播尚不全面了解。本研究采用多方面的方法来监测总需氧细菌以及产生碳青霉烯酶的肠杆菌门 (CPE) 的存在、微生物群和汇排水生物膜 (SDB) 和医院废水 (WW) 的抵抗组在法国的同一医疗机构中。从 2023 年 1 月到 4 月,在格勒诺布尔阿尔卑斯大学医院的新生儿 (NICU) 和成人 (AICU) ICU 中每月收集 SDB 和 WW 样本。在 NICU 中,常规使用表面活性剂进行水槽引流消毒。在 AICU 中,不进行常规消毒。在非选择性培养基上定量可培养的需氧细菌,并使用两种选择性琼脂筛选 CPE。通过 MALDI-TOF MS 鉴定分离株,并对肠杆菌和铜绿假单胞菌进行抗生素敏感性试验 (AST)。通过靶向 >80 ARGs 和 MGEs 的高通量 qPCR 分析电阻组。通过全长 16S rRNA 测序评估总体细菌微生物群。通过细菌培养未从任一 ICU 的 SDB 中分离出 CPE。不依赖培养的方法揭示了两个 ICU 中 SDB 的总体不同的微生物群组成。 AICU SDBs 以含有革兰氏阴性菌属的病原体为主,包括假单胞菌属、窄食单胞菌属、克雷伯菌属和革兰氏阳性葡萄球菌属,而 NICU SDBs 以革兰氏阴性属无色杆菌属、沙雷氏菌属和 Acidovorax 属以及革兰氏阳性属 Weisella 和 Lactiplantibacillus 为主.相比之下,SDBs 的抵抗组在两个 ICU 之间没有表现出显着差异,表明 ARGs 和 MGEs 的丰度与微生物群组成和消毒做法无关。AICU WW 表现出比 NICU WW 更明显的需氧细菌。此外,AICU WW 产生了 15 个 CPE,而 NICU WW 产生了一个 CPE。所有 CPEs 均在物种水平上进行了表征。NICU 和 AICU WW 样本的微生物群与各自的 SDB 不同,并且在四个月期间表现出明显的变化:AICU WW 包含更多赋予对喹诺酮类药物和整合子整合酶基因耐药性的基因,而 NICU WW 表现出较高的链阳蛋白耐药基因丰度。我们的研究表明,所调查的医疗机构的两个 ICU 中医院 SDB 的抵抗组与微生物群、环境和当地消毒措施无关。然而,从调查的排水管中收集废物的 WW 管道中 CPE 的流行率有所不同。这些发现为 ICU 中 SDB 中耐药基因的恢复力提供了有价值的见解,强调了创新策略对抗临床环境中抗菌素耐药性的必要性。
更新日期:2025-06-02
中文翻译:

医院水槽排水生物膜抵抗组独立于相应的微生物群、环境和消毒措施
在医院,抗生素耐药菌 (ARB) 可能通过水槽排水管中的生物膜传播,这可能导致感染。尽管汇引流管在院内感染中 ARB 的传播中具有潜在作用,但大多数医院缺乏对这些引流管的常规监测。因此,目前对 ARB 的传播以及抗菌素耐药基因 (ARG) 和相关的移动遗传元件 (MGE) 通过下水道的传播尚不全面了解。本研究采用多方面的方法来监测总需氧细菌以及产生碳青霉烯酶的肠杆菌门 (CPE) 的存在、微生物群和汇排水生物膜 (SDB) 和医院废水 (WW) 的抵抗组在法国的同一医疗机构中。从 2023 年 1 月到 4 月,在格勒诺布尔阿尔卑斯大学医院的新生儿 (NICU) 和成人 (AICU) ICU 中每月收集 SDB 和 WW 样本。在 NICU 中,常规使用表面活性剂进行水槽引流消毒。在 AICU 中,不进行常规消毒。在非选择性培养基上定量可培养的需氧细菌,并使用两种选择性琼脂筛选 CPE。通过 MALDI-TOF MS 鉴定分离株,并对肠杆菌和铜绿假单胞菌进行抗生素敏感性试验 (AST)。通过靶向 >80 ARGs 和 MGEs 的高通量 qPCR 分析电阻组。通过全长 16S rRNA 测序评估总体细菌微生物群。通过细菌培养未从任一 ICU 的 SDB 中分离出 CPE。不依赖培养的方法揭示了两个 ICU 中 SDB 的总体不同的微生物群组成。 AICU SDBs 以含有革兰氏阴性菌属的病原体为主,包括假单胞菌属、窄食单胞菌属、克雷伯菌属和革兰氏阳性葡萄球菌属,而 NICU SDBs 以革兰氏阴性属无色杆菌属、沙雷氏菌属和 Acidovorax 属以及革兰氏阳性属 Weisella 和 Lactiplantibacillus 为主.相比之下,SDBs 的抵抗组在两个 ICU 之间没有表现出显着差异,表明 ARGs 和 MGEs 的丰度与微生物群组成和消毒做法无关。AICU WW 表现出比 NICU WW 更明显的需氧细菌。此外,AICU WW 产生了 15 个 CPE,而 NICU WW 产生了一个 CPE。所有 CPEs 均在物种水平上进行了表征。NICU 和 AICU WW 样本的微生物群与各自的 SDB 不同,并且在四个月期间表现出明显的变化:AICU WW 包含更多赋予对喹诺酮类药物和整合子整合酶基因耐药性的基因,而 NICU WW 表现出较高的链阳蛋白耐药基因丰度。我们的研究表明,所调查的医疗机构的两个 ICU 中医院 SDB 的抵抗组与微生物群、环境和当地消毒措施无关。然而,从调查的排水管中收集废物的 WW 管道中 CPE 的流行率有所不同。这些发现为 ICU 中 SDB 中耐药基因的恢复力提供了有价值的见解,强调了创新策略对抗临床环境中抗菌素耐药性的必要性。


















































 京公网安备 11010802027423号
京公网安备 11010802027423号